Vergeleken met andere polymeren is DNA eigenlijk een simpel molecuul. DNA is een lange keten samengesteld uit 4 bouwstenen. Deze bouwstenen worden ook wel “basen” of “nucleotiden” genoemd. De bouwstenen van DNA zijn adenine (of A), cytosine (of C), guanine (of G) en thymine (of T).DNA lijkt een willekeurige aaneenschakeling van A’s, C’s, G’s en T’s, maar de volgorde van deze letters vormt in feite de genetische code. Elk blokje van 3 basen, een codon genoemd, codeert voor een aminozuur. Zo codeert bijvoorbeeld TGT voor het aminozuur cysteine, of CGT voor het aminozuur arginine. De 4 basen kunnen 64 verschillende codons vormen. Er zijn echter maar 20 aminozuren (tabel 1). Daarom zijn er meerdere codons die voor hetzelfde aminozuur kunnen coderen (tabel 2).
|
Aminozuur
|
3-letter afkorting
|
1-letter symbool
|
| Alanine |
Ala
|
A
|
| Arginine |
Arg
|
R
|
| Asparagine |
Asn
|
N
|
| Asparaginezuur |
Asp
|
D
|
| Cysteine |
Cys
|
C
|
| Glutamine |
Gln
|
Q
|
| Glutaminezuur |
Glu
|
E
|
| Glycine |
Gly
|
G
|
| Histidine |
His
|
H
|
| Isoleucine |
Ile
|
I
|
| Leucine |
Leu
|
L
|
| Lysine |
Lys
|
K
|
| Methionine |
Met
|
M
|
| Fenylalanine |
Phe
|
F
|
| Proline |
Pro
|
P
|
| Serine |
Ser
|
S
|
| Threonine |
Thr
|
T
|
| Tryptofaan |
Trp
|
W
|
| Tyrosine |
Tyr
|
Y
|
| Valine |
Val
|
V
|
Tabel 1: de aminozuren met hun symbolen
|
Eerste Positie
|
Tweede Positie
|
Derde Positie
|
|||
|
T
|
C
|
A
|
G
|
|
|
|
T
|
Phe
Phe Leu Leu |
Ser
Ser Ser Ser |
Tyr
Tyr Stop Stop |
Cys
Cys Stop Trp |
T
C A G |
|
C
|
Leu
Leu Leu Leu |
Pro
Pro Pro Pro |
His
His Gln Gln |
Arg
Arg Arg Arg |
T
C A G |
|
A
|
Ile
Ile Ile Met |
Thr
Thr Thr Thr |
Asn
Asn Lys Lys |
Ser
Ser Arg Arg |
T
C A G |
|
G
|
Val
Val Val Val |
Ala
Ala Ala Ala |
Asp
Asp Glu Glu |
Gly
Gly Gly Gly |
T
C A G |
Tabel 2: de genetische code
Expressie van de erfelijke code: transcriptie, splicing en translatie
De erfelijke code, bepaald door de volgorde van de basen, is vast opgeslagen in het DNA. Het DNA ligt op z’n beurt weer opgeslagen in de chromosomen, in de kern van elke cel (figuur 1). Het tot expressie brengen van de erfelijke code gebeurt in 3 stappen. Allereerst wordt de genetische informatie buiten de kern gebracht nadat het DNA is overgeschreven in een andere vorm, het RNA. Dit proces van overschrijven heet transcriptie.
Eenmaal buiten de kern ondergaat het RNA een bewerking (de tweede stap) waarbij niet-relevante (niet-coderende) stukken uit het RNA geknipt worden. Dit proces heet splicing . Na de splicing blijft het functionele messenger RNA (mRNA) over. Het mRNA dient nu als een soort matrijs waarop een eiwit geproduceert wordt: translatie van RNA in eiwit (de derde stap). Voor een correct verloop van transcriptie, splicing en translatie is het noodzakelijk dat er in de erfelijke code signalen liggen opgeslagen die aangeven waar een gen begint, wat coderend materiaal is en waar de vertaling naar eiwit begint en moet ophouden (figuur 2).
Deze signalen bestaan in de vorm van een aantal specifieke codons of een reeks basen in specifieke volgorde. De zogenaamde splice sites, bestaande uit een unieke serie van 6 basen, geven aan waar een serie codons begint (die uiteindelijk in een keten van aminozuren wordt vertaald) en waar die ophoudt. De splice sites verdelen daarmee een gen in coderende stukken, exonen genoemd, en in niet-coderende stukken, intronen genoemd. Hier kunt u een in korte animatie bekijken hoe de splicing in z’n werk gaat.
Voor de translatie zijn er een aantal codons die van essentieel belang zijn. Het codon ATG vormt een startsignaal. Het startcodon ATG codeert tevens voor het aminozuur methionine en vandaar dat alle eiwitten in eerste instantie met methionine beginnen. Vaak wordt dit bij verdere rijping van een eiwit weer afgesplitst. Verder zijn er 3 codons die een eindsignaal vormen: de codons TAA, TGA en TAG worden de stopcodons genoemd (tabel 2). Een gen, een DNA fragment dus dat de code voor een eiwit bevat, begint met een startcodon en eindigt met een stopcodon.
In het zeer aan te bevelen boek “Genome, the autobiography of a species in 23 chapters” maakt de auteur Matt Ridley een prachtige vergelijking tussen het totaal aan genen, het genoom, en een boek:
“Stelt u zich voor dat het genoom een boek is.
Het boek bevat 23 hoofdstukken die chromosomen heten.
Elk hoofdstuk bevat duizenden verhalen die we genen noemen.
Elk verhaal bestaat uit paragrafen, exonen genoemd, die worden onderbroken door advertenties, intronen genoemd.
Elke paragraaf wordt gevormd door woorden, die codons heten,
en elk woord wordt geschreven in letters die we basen noemen.
Het boek bevat 2 miljard woorden….even dik als 800 Bijbels.
….Het is een gigantisch document, een immens boek, een recept van extravagante omvang, en dat past allemaal in de microscopisch kleine kern van een cel, die kleiner is dan een speldepunt.”
Het is overigens aardig te bedenken dat de organisatie en verwerking van informatie op de harde schijf van een computer of op een CD goed te vergelijken is met de organisatie en verwerking van de erfelijke code.
Mutaties
Hoe mooi de erfelijke informatie ook georganiseerd is, er kunnen veel dingen misgaan. Hierbij doelen we op mutaties: veranderingen van het erfelijk materiaal waardoor eiwitten niet meer goed werken of zelfs niet meer gemaakt worden.
Op het RNA worden de codons afgelezen en letterlijk vertaald in aminozuren die aan elkaar worden gezet: de translatie. Een onjuiste code heeft dan direct gevolg voor de productie of de werking van het eiwit.
Indien in bovenstaand voorbeeld de eerste T in het TGT codon voor cysteine zou veranderen in een C, dan ontstaat het codon CGT voor arginine. In dit geval wordt er dus in plaats van een cysteine een arginine in het eiwit in gebouwd. Dit kan verstrekkende gevolgen hebben. Cysteine is een aminozuur dat in een eiwit dwarsverbindingen maakt en zo zorgt voor een functionele driedimensionale structuur. Zo kunnen veranderingen van aminozuren meer of minder ernstige gevolgen voor het functioneren van een eiwit hebben, afhankelijk van de aard van de aminozuurverandering of de plaats in het eiwit waar de verandering is opgetreden.
Het gebeurt regelmatig dat een codon verandert terwijl de code voor het aminozuur hetzelfde blijft. Zoals uit tabel 2 blijkt zijn er meerdere codons die voor hetzelfde aminozuur coderen. In zo’n geval spreken we vrijwel altijd van een neutrale variant: de verandering in het DNA leidt niet tot een klinisch effect, in dit geval hypercholesterolemie. Dergelijke neutrale varianten worden ook veelvuldig gevonden bij mensen met een normaal cholesterolgehalte.
Een andere mogelijkheid is dat door een of andere oorzaak een extra base in een gen wordt gezet, of dat er een verdwijnt. De veelvoud van 3 is dan weg en in eerste instantie worden er verkeerde codons afgelezen, totdat er door de verandering een stopcodon ontstaat. Dit zijn ernstige zogenaamde “frame-shift” mutaties die er meestal toe leiden dat er geen eiwit wordt gemaakt. Deze mutaties heten ”nul-mutaties” of “nul-allelen”.
Een verandering van de basenvolgorde kan ook gevolgen hebben voor de transcriptie- en translatie-signalen. Als het codon TCG voor serine verandert in TAG, dan wordt dit een stopcodon waardoor de transcriptie voortijdig wordt beëindigd.
Veranderingen in de splice sites leiden tot het onterecht uitknippen van exonen of tot het onterecht vertalen van intronen: kleine veranderingen in de basenvolgorde met grote veranderingen in het eiwit.
Tenslotte noemen we nog de grote gen-rearrangementen: mutaties waarbij enkele honderden of zelfs duizenden basen uit het gen verdwenen zijn (deleties) of er juist zijn ingezet (inserties). Uiteraard zijn dit veranderingen die funest zijn voor werking van het eiwit.
Het LDL-receptor gen en mutaties
Het complete LDL-receptor gen bestaat uit ongeveer 45000 basen (of 45 kilobasen(kb)), verdeeld over 18 exonen en 17 intronen (figuur 3). Na de transcriptie, het overschrijven van DNA in RNA blijft er een mRNA over van circa 2500 basen. Minder dan 6% van het LDL-receptor gen is maar coderend materiaal.

Figuur 3: het LDL-receptor gen
Het LDL-receptor gen is evolutionair gezien een zeer oud gen. Het menselijk LDL-receptor gen komt voor 80% in basenvolgorde overeen met de kikker Xenopus laevis.
Mens en kikker besloten 350 miljoen jaar geleden uit elkaar te gaan, waarmee het LDL-receptor gen behoorlijk oud moet zijn. Tijd genoeg voor dit gen om mutaties te verzamelen. Wereldwijd zijn er al meer dan 900 verschillende beschreven. Dit heeft zeker te maken met het feit dat FH geen dodelijke aandoening is op jonge leeftijd. Dragers van een mutatie krijgen volop de gelegenheid deze aan hun nakomelingen door te geven.
De nomenclatuur van mutaties is een verhaal apart. In het verleden was het een traditie om een mutatie de naam te geven van de stad of het land waar de eerste patiënt met die mutatie vandaan kwam. Zo kennen we FH-Amsterdam (gevonden in Amerika, in een patient van Nederlandse afkomst), FH-Piscataway, FH-Vancouver-6 of FH-Germany. Met de grote aantallen mutaties was deze benaming niet meer praktisch en werd er gekozen voor een meer functionele nomenclatuur.
De meeste mutaties worden aangegeven door de aminozuurverandering en het nummer van het veranderde aminozuur te noemen. Bijvoorbeeld V408M is een mutatie waarbij het 408-ste aminozuur valine (V) is veranderd in methionine (M).
in het gen:
~~~~AGG AAC GTG GTC GCT~~~~muteert naar~~~~AGG AAC ATG GTC GCT~~~~
positie 1285 in het 408-ste codon
in het eiwit:
~~~~arg-asn-val-val-ala~~~~ wordt dan ~~~~arg-asn-met-val-ala~~~~
408-ste aminozuur
Bij de W23X-mutatie is op de plaats van het 23-ste aminozuur tryptofaan (W) een stopcodon (X) ontstaan.
in het gen:
~~~~TAC AAG TGG GTC TGC~~~~muteert naar~~~~TAC AAG TAG GTC TGC~~~~
positie 131 in het 23-ste codon => stopcodon (TAG)
gevolg: er wordt geen volwaardig mRNA gemaakt en dus ook geen eiwit.
Helaas zijn niet alle mutaties op deze eenvoudige wijze aan te geven. Met name als het niet een aminozuur verandering betreft wordt het ingewikkeld. In plaats van het nummer van het aminozuur kunnen we ook het nummer van de betreffende base aangeven: 600delC betekent dat de 600-ste base C verdwenen is. 1645insC betekent dat achter de 1645-ste base een extra C is ingevoegd.
frame shift mutatie door een deletie:
~~~~GCC TTC GAG TTC CAC TGC CTA AGT~~~~
C op positie 600 verdwijnt en de codons schuiven 1 positie naar links
~~~~GCC TTG AGT TCC ACT GCC TAA GT ~~~~
6 codons verderop ontstaat een stopcodon (TAA)
Nu komt de volgende complicatie. Omdat bij de opheldering van de functie en structuur van genen vroeger alleen aan exonen aandacht werd besteed, zijn de basen van de intronen nooit genummerd. Als we nu aan een bepaalde base in een intron willen refereren, dan kan dit alleen door van de dichtstbijzijnde genummerde base van een exon uit te gaan. Bijvoorbeeld, bij de 1358+1 mutatie is na de laatste base van exon 9 op positie 1358 de eerste base van intron 9 van een G naar een A veranderd.
exon 9~~~~AAT GAT CTG CAG gtg agc~~~~intron 9~~~~cct cag CAC CCA~~~~exon 10
laatste base in exon 9: 1358 1358+1 1359-1 1359 eerste base in exon 10
Op analoge wijze is bij de 1359-1 mutatie de laatste base G van intron 9, voor de eerste base op positie 1359 van exon 10, veranderd in een A. Bij 191-2 is de voorlaatste base A in intron 2, voor positie 191 van de eerste base van exon 3, veranderd in een G.
Tenslotte de grote deleties in inserties. Omdat in de meeste gevallen niet tot in detail bekend is waar grote stukken gen zijn weggevallen of zijn ingezet en deze mutaties relatief zeldzaam zijn, beperkt men zich tot het gebruik van traditionele namen zoals boven.
Voorbeelden zijn FH-Cape Town-2, wat een deletie is van 2500 basen waarbij exonen 7 en 8 (en gedeelten van intronen 6 tot en met 8 ) zijn verdwenen, of FH-Alkmaar, wat een insertie is van exonen 3 tot en met 8 (inclusief stukjes van intron 2 en intron 8 aan de uiteinden) in intron 8.

FH-Cape Town-2: 2500 basen van exon 7 en 8 zijn verdwenen.
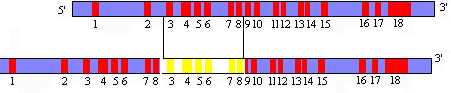
FH-Alkmaar-1: 10000 basen van exon 3 tot en met 8 zijn in intron 8 gezet.